DNA en de eiwitsynthese
Filmpje: Het leven binnen in een cel
"The inner life of the cell" door Harvard University
Bekijk in dit YouTube filmpje hoe de systemen binnen een cel werken (o.a. de organellen nodig bij de eiwitsynthese).
Je herkent de volgende dingen achter elkaar:
- Een bloedstroom in een bloedvat. Langs de wanden wandelen witte bloedcellen.
- Een soort soep, dat is het celmembraan (met dubbele laag) en daarna de rand van de cel. Er drijven verschillende transporteiwitten in, die er doorheen steken.
- Het celskelet en celspieren: opbouwen en afbreken van de tentstokken, waardoor de cel van vorm kan veranderen.
- Het verplaatsen van een transportblaasje, het loopt met pootjes langs een stok van het celskelet.
- De lange rupsen in de cel zijn de mitochondriën.
- De kern met gaatjes waar DNA-draden doorheen komen. Daar worden ze als een rits gelezen door ribosomen (bestaan uit 2 gele brokjes), die erna een eiwitketting uit poepen. De eiwitketen gaat daarna direct uit zichzelf opvouwen.
- Wat op een lavalamp lijkt is het golgi-systeem.
- De blaasjes (lysosomen) die van het golgi-systeem worden afgescheiden, worden naar het celmembraan gebracht (met de pootjes langs de stok) en de stoffen erin worden naar buiten afgescheiden.
LINK WERKT NIET??? Noot: Harvard haalt regelmatig het filmpje weer van YouTube af (gesteggel met auteursrechten?), maar als je op YouTube zoekt naar "The inner life of the cell" dan vind je het filmpje wel weer (duurt ong. 8 min.).
Eiwitsynthese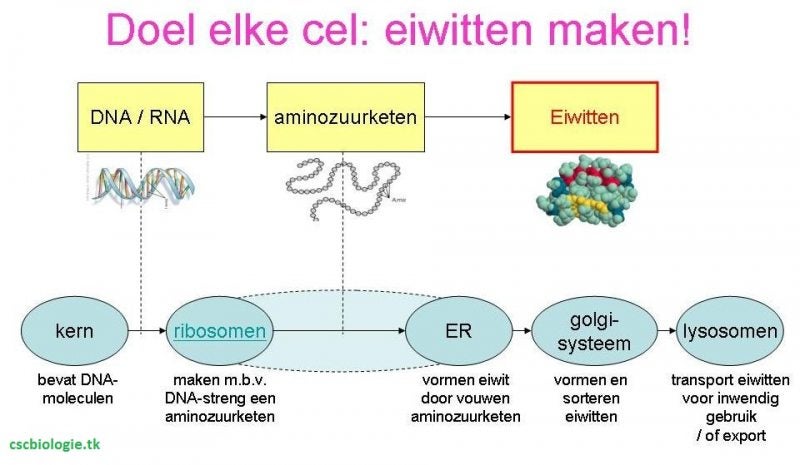

DNA-synthese / -replicatie / -verdubbeling
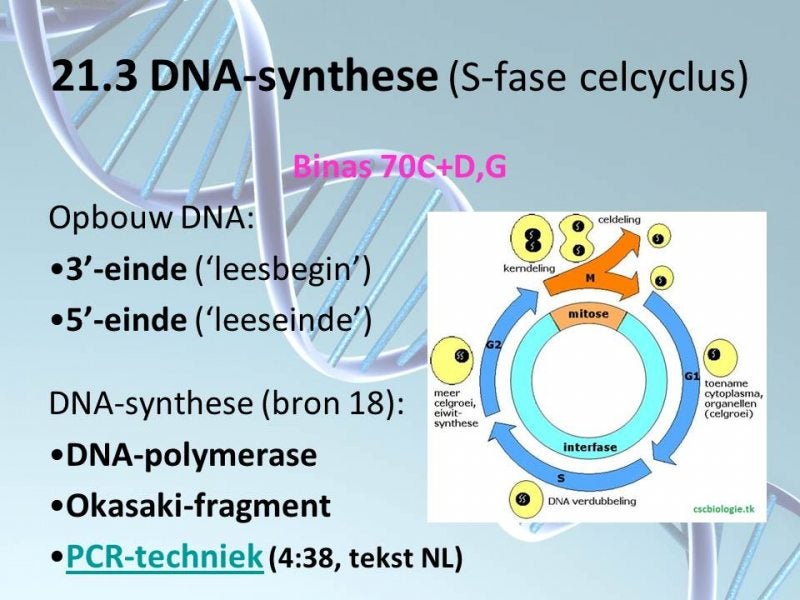
DNA-synthese in de cel vindt plaats tijdens de S-fase van de celcyclus (zie plaatje hieronder).
Celcyclus met mitose

Filmpje genexpressie (2:20 min.)
Noot: Het filmpje wil niet op iedere pc werken! Ik ben er nog niet achter gekomen waarom. (Als na het klikken het eerste beeld van het filmpje niet verschijnt, doet hij het niet.)
Hoe je het filmpje moet bekijken:
Niet alles wat er te zien is, moet je begrijpen. Sommige dingen krijg je pas in 6V (zoals introns en exons=selecteren van de nodige stukjes mRNA). Kijk naar de plaatjes en probeer wat dingen te herkennen. Deze kunnen je helpen bij de stof over hoe de expressie (het werken) van genen gaat.
Link naar filmpje: klik op het plaatje
Variatie in DNA
Variatie in erfelijk materiaal:



PCR-methode: zie Binas 70D.
Filmpje (voor de Nederlandse vertaling, zie hieronder!)
(Begrippen: PCR, primer, DNA-polymerase)
Tekst NL bij PCR film
Begin film
Met PCR of Polymerase Chain Reaction, wat staat voor Polymerase ketting reactie kan men heel specifieke stukken DNA selectief vermeerderen in een complexe mix van DNA moleculen.
…. Korte pauze….
Voorbeeld
In dit voorbeeld wordt het volledige DNA weergegeven in het blauw. Daarop bevindt zich het stuk DNA dat we willen vermeerderen. Dit stuk is aangeduid in het groen en we noemen dit de doelsequentie of target sequence.
Met PCR kunnen we miljoenen kopieën van de target sequence maken zonder het te moeten opzuiveren.
Begin cycle 1
In de eerste cyclus van de PCR reactie, worden de twee DNA strengen van elkaar gescheiden door de temperatuur te doen stijgen tot 95°C. Als de twee DNA strengen gescheiden zijn wordt de temperatuur verlaagd tot 60°C zodat een paar specifieke ontworpen primers zich kunnen vasthechten.
Één primer op elke streng van het DNA.
Daarna wordt de temperatuur weer verhoogd tot 72°C zodat het polymerase enzym op de primers kan gaan zitten en vervolgens de vrije nucleotiden uit de oplossing kan halen om zo de primers verlengen.
In het hele mengsel van DNA moleculen worden alleen de stukken DNA waarop de primers zaten verlengd. Aan het einde van cyclus 1 is onze DNA streng verlengd tot twee gedeeltelijk overlappende moleculen dubbelstreng DNA.
Begin cylcle 2
In de volgende cyclus worden dezelfde stappen herhaald. Eerst worden de DNA strengen van elkaar gehaald bij 95°C. Daarom is het zo nuttig dat het polymerase warmte stabiel is, zo wordt het niet gedeactiveerd dij deze hoge temperatuur wat wel zou gebeuren bij andere enzymen. Bij 60°C gaan de primers op de DNA strengen zitten. En het polymerase verlengt de strengen als de temperatuur op 72° wordt gebracht.
Na cyclus 2 zijn er al vier keer zoveel kopieën DNA die de target sequence bevatten.
Begin cylce 3
In de derde cyclus haalt men de DNA strengen weer van elkaar,….
de primers gaan vastzitten ,….
en worden verlengt.
Cyclus 3 produceert twee stukken dubbelstreng DNA die alléén de target sequence bevatten; ook de target moleculen genoemd en 6 DNA moleculen die daarnaast ook langere stukken bevatten.
Het is nu de bedoeling om de DNA moleculen aan de rechterkant zoveel mogelijk te kopiëren.
Begin cycle 4
In de vierde cyclus gebeurt weer net hetzelfde;
De stengen worden gescheiden bij 95°……
De primers gaan vastzitten bij 60° ……
En het polymerase verlengt ze bij 72°…
Aan het einde van cyclus vier zijn er 8 target moleculen en 8 moleculen met langere stukken nucleotiden.
Begin cycle 5
Net hetzelfde gebeurt er in de vijfde cyclus;
De stengen worden gescheiden ……
De primers gaan vastzitten ….
En de primers worden verlengd …
Aan het einde van cyclus vijf zijn er 22 target moleculen en maar 10 moleculen met langere stukken nucleotiden.
… by the end of cycle 30 ...
Het aantal target moleculen zal exponentieel toenemen bij elke cyclus en aan het einde van cyclus 30, zal het aantal zijn opgelopen tot meer dan een miljard kopieën. Terwijl er maar 60 kopieën zijn van moleculen met langere stukken.
We kunnen nu besluiten dat de oplossing bijna alleen maar bestaat uit target sequence DNA moleculen.
En dit DNA kunnen we nu gebruiken om verder onderzoek op te doen.
DNA-synthese (verdubbeling van DNA)
Antwoorden van petje op/petje af - vragen in de les
De verdubbeling van DNA (DNA-synthese) is te vinden in Binas tabel 70C3.
(PCR staat trouwens in 70D.)
DNA-synthese vindt plaats alvorens een kerndeling (mitose of meiose). Dat gebeurt tijdens de S-fase van de celcyclus (zie plaatje hierboven).
Okasaki-fragmenten zijn korte stukjes DNA die ontstaan bij de achterwaartse synthese van de coderende streng.
(Strikvraag! Zie Binas 70C3 samen met 70G. Hier zie je dat de streng die bij 3' begint, de matrijsstreng is. Maar PAS OP! Want 70G geeft niet de DNA-synthese weer maar de eiwitsynthese! Het gaat me hier alleen om het onderscheiden van de matrijsstreng en de coderende steng!).
In dubbelstrengs DNA zijn de percentages A- en T-basen aan elkaar gelijk. (Want tegenover elke A staat een T en andersom.)
In elk stuk DNA zijn de percentages A- en T-basen niet aan elkaar gelijk.
(Want in een willekeurig stuk (enkelstrengs) DNA is de verdeling A, T, C en G steeds verschillende door de aanwezigheid van verschillende genen/codes.)
Functies van: LET OP! We hebben het nu alleen over DNA-synthese, géén eiwitsynthese! Wat ik heb onderstreept, is heel verwarrend met de eiwitsynthese.
- RNA-primer: startpunt vorming nieuwe DNA-strengen
- DNA-polymerase: verbindt nucleotiden
- DNA-ligase: verbindt DNA-fragmenten/Okasaki-fragmenten tijdens achterwaartse synthese
- Helicase: verbreekt H-bruggen (=bruine driehoekje in bron 18 op p56)
Eiwitsynthese voor gevorderden
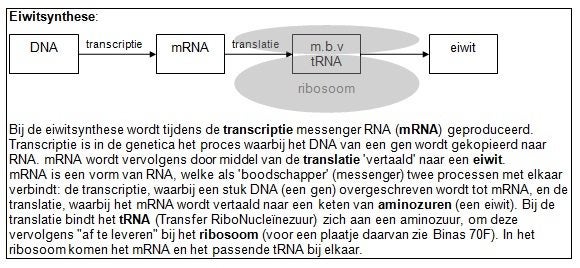
Filmpje DNA Transcription and Protein Assembly; Eiwitsynthese voor gevorderden
DNA Translatie in de ribosoom, van aflezen mRNA → eiwitketen m.b.v. codons, anticodons, tRNA en aminozuren. (=Still uit filmpje op 1:53)
! Vergelijk dit filmpje + plaatje met Binas 70G en 70I en herken!!!

Functies van: LET OP! We hebben het nu over de eiwitsynthese, géén DNA-synthese! Wat ik heb onderstreept, is heel verwarrend met de DNA-synthese (21.3).
- RNA-polymerase (TAC) = splitst DNA-strengen
- mRNA = messenger-RNA
- tRNA = transfer-RNA
- rRNA = ribosomaal RNA (deel van ribosoom/enzym)

Codon = 3 ‘letters’ (basen) achter elkaar die een teken geven. Ze staan in Binas 70E.
Bijv. startcodon mRNA = AUG, stopcodons = UAA, UGA, UAG.
Repressoreiwit = eiwit dat voor startplaats aan DNA bindt à transcriptie geblokkeerd.
Inductoreiwit = laat repressoreiwit loslaten, daardoor transcriptie weer mogelijk.